Le fonctionnement des êtres vivants est assuré par un ensemble de réactions physico-chimiques catalysées par des protéines appelées les enzymes. Les enzymes sont synthétisées dans les cellules en fonction des besoins de l'organisme. L'information nécessaire à cette synthèse ainsi qu'à celle des protéines structurales est stockée dans chaque cellule au sein de macromolécules d'Acide DésoxyriboNucléique (ADN).
L'Académie Royale des Sciences de Suède a décerné le prix Nobel de chimie 2006 à Roger D. Kornberg, chercheur américain à l'université de Stanford, « pour ses travaux sur les bases moléculaires de la transcription des gènes chez les eucaryotes » [1]. Cet article détaille la nature des travaux récompensés après un rappel des différentes étapes de la transcription des gènes.
Le fonctionnement des êtres vivants est assuré par un ensemble de réactions physico-chimiques catalysées par des protéines appelées les enzymes [2,3]. Les enzymes sont synthétisées dans les cellules en fonction des besoins de l'organisme. L'information nécessaire à cette synthèse ainsi qu'à celle des protéines structurales est stockée dans chaque cellule au sein de macromolécules d'Acide DésoxyriboNucléique (ADN).
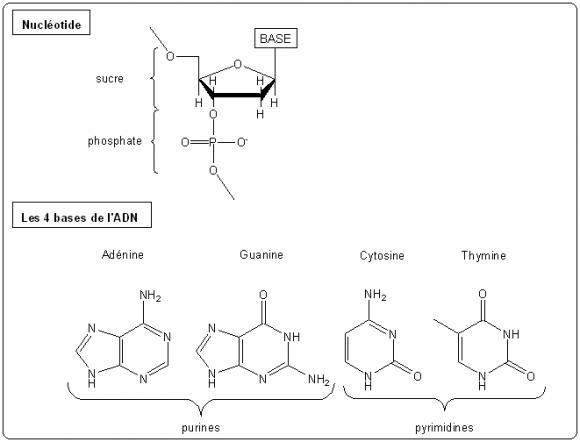
La structure de l'ADN a été élucidée en 1953 par Watson et Crick[1], c'est un polymère de nucléotides , composés chacun de trois parties (figure 1) :
-
un groupement phosphate
-
un sucre désoxyribose à 5 atomes de carbone
-
une base azotée parmi les quatre suivantes : l'adénine (A), la guanine (G), la thymine (T) et la cytosine (C).
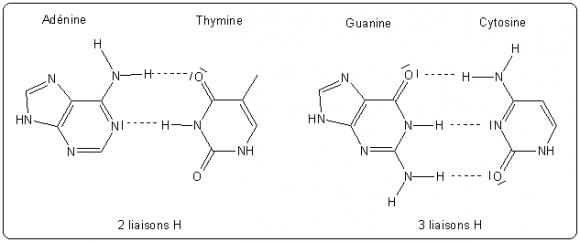
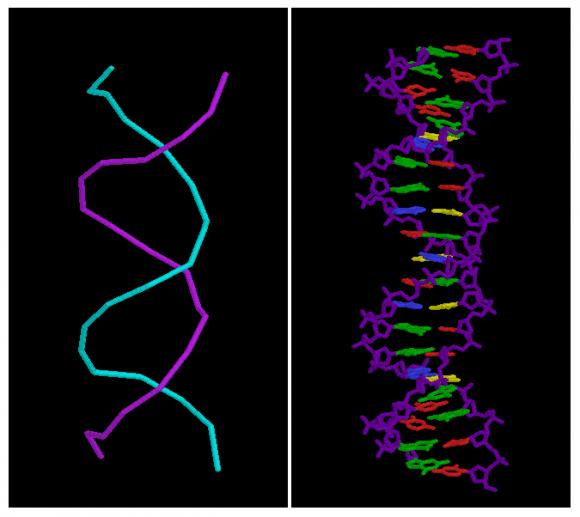
Les bases A-T et G-C sont complémentaires et l'ADN est en fait formé de deux brins de polymères dans lesquels chaque base d'un brin est en interaction avec son complémentaire par liaisons hydrogène (figure 2). Le double brin présente une structure hélicoïdale (figure 3), on parle de « structure en double hélice » pour l'ADN [4]. L'ordre dans lequel les bases sont enchaînées dans l'ADN constitue le code de l'information génétique : chaque protéine est codée par une séquence particulière appelée un gène.
Les biologistes divisent le vivant en deux grandes familles d'organismes en fonction de l'existence ou non d'un noyau cellulaire dans lequel est confiné l'ADN. Ils distinguent ainsi :
-
les procaryotes qui correspondent aux bactéries. Ce sont des organismes monocellulaires au sein desquels l'ADN n'est pas séparé du cytoplasme (milieu cellulaire).
-
les eucaryotes, organismes mono ou pluricellulaires au sein desquels l'ADN est confiné dans un noyau séparé du cytoplasme par une double membrane. Les êtres humains font partie de cette deuxième catégorie.
Dans le cas des eucaryotes, l'ADN ne quitte jamais le noyau. Or les ribosomes, qui sont les organites qui synthétisent les enzymes, sont situés dans le cytoplasme, à l'extérieur du noyau. Il faut donc transférer l'information, depuis le noyau vers le cytoplasme. C'est le rôle de l'acide ribonucléique (ARN), copie du gène, qui va quitter le noyau pour rejoindre les ribosomes où il sera traduit en enzyme. La synthèse de l'ARN à partir de l'ADN au sein du noyau est appelée la transcription.
La transcription de l'ADN en ARN se déroule selon les étapes suivantes :
-
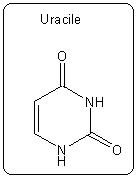
L'initiation : la séquence des bases à transcrire est indiquée par une zone d'ADN appelée « promoteur ». Une fois le promoteur reconnu, une enzyme, l'ARN-polymérase, se fixe sur l'ADN dont les deux brins se séparent localement. Les nucléotides libres du milieu nucléaire vont pouvoir venir s'apparier avec leur complémentaire sur le brin support de la transcription à ceci près que la thymine est remplacée par une base de structure très proche : l'uracile (figure 4), également complémentaire à l'adénine. La polymérase catalyse la jonction entre ces nucléotides qui forment le brin d'ARN.
-
L'élongation : l'enzyme se déplace le long de l'ADN en séparant les deux brins sur son passage. L'appariement des nucléotides se poursuit, et la chaîne d'ARN formée se détache progressivement de l'ADN ce qui permet aux deux brins de s'apparier à nouveau après le passage de la polymérase.
-
La terminaison : l'enzyme rencontre une zone d'ADN appelée « terminateur » qui signale l'arrêt de la transcription. L'enzyme et le brin d'ARN se décrochent de l'ADN qui retrouve sa structure initiale. L'ARN commence alors une période de maturation pendant laquelle il subit une série de transformations avant de rejoindre les ribosomes où commencera la synthèse enzymatique. On peut noter que le brin d'ARN est formé par complémentarité avec le brin d'ADN qui sert de support à la transcription, c'est donc une copie de l'autre brin d'ADN avec un remplacement des bases thymines par des uraciles.
Ce schéma n'est qu'une version simplifiée de ce qui se déroule réellement, la polymérase agit en fait conjointement à d'autres protéines indispensables à la transcription [2,5]. Cinq d'entre elles sont appelées les « cofacteurs de transcription », elles reconnaissent le promoteur et s'y fixent puis forment un complexe avec la polymérase. Il intervient également des protéines activatrices ou inhibitrices de la polymérase ainsi qu'un complexe d'une vingtaine de protéines appelé le médiateur qui permet de transmettre les informations de régulation depuis les inhibiteurs ou activateurs jusqu'à la polymérase. Le grand nombre des protéines mises en jeu rend le processus de transcription très complexe mais en permet une régulation très fine.
La contribution pour laquelle Kornberg est plus particulièrement récompensé est l'obtention de structures cristallographiques de la machinerie de transcription en pleine action.
La cristallographie est une technique qui permet d'accéder à la structure microscopique de la matière grâce à l'interprétation de la figure de diffraction des rayons X par le réseau cristallin.
En 2001, Kornberg et ses collaborateurs résolvent la structure d'une ARN-polymérase de levure, organisme eucaryote au sein duquel cette enzyme présente des analogies importantes avec la forme humaine [6]. La résolution est de 0,28 nm (2,8 Å), ce qui permet un niveau de détail très élevé : on parle de « résolution atomique » puisque le rayon des atomes est de l'ordre de 0,1 nm. Les premières structures décrites montrent que la polymérase sépare effectivement les deux brins d'ADN et maintient le brin support dans une position particulière. Il se forme alors en face du nucléotide de l'ADN à transcrire une minuscule cavité parfaitement adaptée à son nucléotide complémentaire et uniquement à celui-ci (cf figure 2). Cela explique la très grande sélectivité du processus d'appariement des bases, sélectivité indispensable pour limiter les erreurs de transcription qui sont à l'origine de cancers et d'autres maladies graves si leur nombre dépasse une sur dix mille.
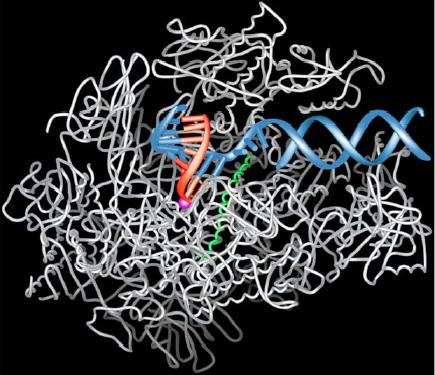
Figure 5. Processus de transcription. Structure de l'ARN-polymerase. L'enzyme est en blanc, la double hélice d'ADN en bleu et l'ARN en cours de formation en rouge. La structure en forme de ressort en vert permet de faire avancer le brin d'ADN dans l'enzyme. Image : communiqué de presse de la fondation Nobel.
Ces travaux sont révolutionnaires car Kornberg a trouvé un moyen de geler la transcription en cours de réalisation. Pour y arriver, il a omis d'introduire une substance qui n'intervient qu'au milieu du mécanisme mais qui est indispensable à son bon déroulement. Le système se fige alors en cours de transcription ce qui laisse le temps de le purifier par une chromatographie d'adsorption puis de le cristalliser afin d'accéder à sa structure par diffraction des rayons X. Avant ces travaux, seules les structures des molécules de départ ou en fin de processus étaient accessibles. Le déroulement de la transcription à l'échelle moléculaire était seulement formulé sous forme d'hypothèses. La technique développée par Kornberg et son équipe a permis d'obtenir une douzaine d'image de la transcription à différents stades, permettant ainsi la première interprétation dynamique du processus.
[1] Le site des prix Nobel (en anglais) : prix Nobel de chimie 2006.
[2] Ouvrages de référence en biochimie : a) D.Voet, J. G. Voet « Biochimie » 2ème éd. 2005, De Boeck. b) J-H. Weil « Biochimie générale » 10ème éd. 2005, Dunod.
[3] Diaporama sur les enzymes en ligne sur CultureSciences-Chimie « Comment marchent les enzymes ? », D. Laage.
[4] « La découverte de la structure de l'ADN », traduction de l'article historique de Watson et Crick dans lequel ils décrivent la structure en double hélice, G. Furelaud, publié sur le site Planet-Vie,
[5] « La transcription chez les eucaryotes », F. Ibarrondo, G. Camus, publié sur le site Planet-Vie
[6] Cramer, P., Bushnell, D.A., Kornberg, R.D. (2001) « Structural basis of transcription: RNA polymerase II at 2.8 ångstrom resolution » Science 292, 1863-1876.
Note
[1] Francis Harry Compton Crick et James Dewey Watson : prix Nobel de physiologie-médecine « pour leurs découvertes concernant la structure moléculaire des acides nucléiques et leur rôle dans le transfert d'information au sein des êtres vivants » en 1962.






